TIANSeq 단일 인덱스 어댑터(Illumina)
특징
■ 편리성: SetA와 SetB는 모두 24개의 단일 인덱스를 제공하며 요구 사항에 따라 자유롭게 선택할 수 있습니다.
■ 간편한 사용: 키트에는 안정성이 강하고 어댑터 용액 희석에 직접 사용할 수 있는 Adapter Dilution Buffer가 장착되어 있습니다.
■ 품질 관리: 엄격한 품질 관리 및 배치 간의 기능 검증을 통해 인덱스 시퀀스의 정확성을 보장합니다.
애플리케이션
1. 이 제품은 NGS(next generation sequencing) 응용 분야에서 Illumina high-throughput sequencing 플랫폼을 위한 DNA 및 RNA 라이브러리 구축에 사용됩니다.
2. 제품의 특정 응용 프로그램에는 전체 엑손 시퀀싱, 표적 시퀀싱, RNA-Seq, ChIP-Seq, 지정 시퀀싱 및 전체 게놈 시퀀싱이 포함됩니다.
3. 이 제품은 methylation 관련 sequencing에 적합하지 않습니다.
어댑터 시퀀스 정보
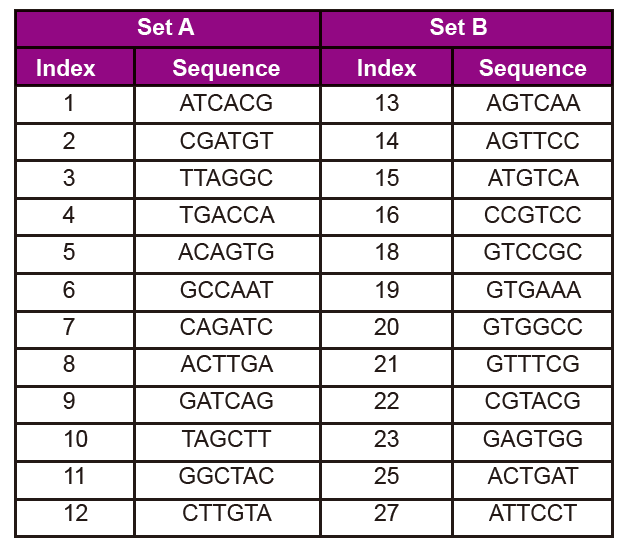
어댑터 시퀀스에는 다음 정보가 포함됩니다.
1. 유니버설 시퀀스
5'-AATGATAACGGCGACCACCGAGATCTACACTCTTTCCCT
ACACGACGCTCTTCCGATCT-3'
2. 시퀀스를 포함한 인덱스
5'-GATCGGAAGAGCACACGTCTGAACTCCAGTCAC[색인1-27]ATCTCGTATGCCGTCTTCTGCTTG-3'
3. 색인 번호 및 순서
모든 제품은 ODM/OEM용으로 사용자 정의할 수 있습니다. 자세한 내용은맞춤형 서비스(ODM/OEM)를 클릭하십시오.
현재 고처리량 시퀀싱 기술은 주로 차세대 시퀀싱 기술을 기반으로 합니다. 차세대 시퀀싱 기술의 읽기 길이는 제한적이므로 전체 길이의 시퀀스를 작은 단편 라이브러리로 분해하여 시퀀싱해야 합니다. 다양한 시퀀싱 실험의 필요에 따라 일반적으로 단일 종단 시퀀싱 또는 이중 종단 시퀀싱을 선택합니다. 현재 차세대 시퀀싱 라이브러리의 DNA 단편은 일반적으로 200-800 bp 범위에 분포되어 있습니다.

a) DNA는 품질이 좋지 않고 억제제가 포함되어 있습니다. 효소 활성의 억제를 피하기 위해 고품질 DNA 샘플을 사용하십시오.
b) PCR-free 방법을 사용하여 DNA 라이브러리를 구축할 때 DNA 샘플의 양이 부족합니다. 단편화된 DNA의 입력이 50ng를 초과하는 경우 라이브러리 구축 과정에서 선택적으로 PCR 없는 워크플로우를 수행할 수 있습니다. Library의 copy number가 너무 낮아서 직접 sequence를 하기 어려운 경우에는 adapter ligation 후 PCR로 DNA library를 증폭할 수 있습니다.
c) RNA 오염은 부정확한 초기 DNA 정량화로 이어집니다. RNA 오염은 게놈 DNA의 정제 과정에서 존재할 수 있으며, 이는 라이브러리 구축 중 부정확한 DNA 정량화 및 불충분한 DNA 로딩으로 이어질 수 있습니다. RNA는 RNase로 처리하여 제거할 수 있습니다.
A-1
a) 작은 조각(60bp-120bp)이 나타납니다. 작은 조각은 일반적으로 어댑터에 의해 형성된 어댑터 조각 또는 이량체입니다. Agencourt AMPure XP 마그네틱 비드를 사용한 정제는 이러한 어댑터 조각을 효과적으로 제거하고 시퀀싱 품질을 보장할 수 있습니다.
b) PCR 증폭 후 라이브러리에 큰 조각이 나타납니다. 어댑터가 연결되면 라이브러리 DNA 조각의 크기가 120bp 증가합니다. Adaptor ligation 후 DNA fragment가 120bp 이상 증가하는 경우 과도한 PCR 증폭으로 인한 fragment 증폭 이상일 수 있습니다. PCR 주기 수를 줄이면 상황을 예방할 수 있습니다.
c) Adapter ligation 후 library DNA fragments의 비정상적인 크기 이 키트의 어댑터 길이는 60 bp입니다. 단편의 두 끝이 어댑터에 연결되면 길이는 120bp만 증가합니다. 이 키트에서 제공하지 않는 어댑터를 사용할 경우 어댑터 길이와 같은 관련 정보를 제공하기 위해 공급업체에 문의하십시오. 실험 워크플로 및 작업이 설명서에 설명된 단계를 따르도록 하십시오.
d) Adapter ligation 전 DNA fragment size가 비정상적일 경우 이 문제의 원인은 DNA fragmentation 시 잘못된 반응 조건으로 인해 발생할 수 있습니다. 다른 DNA 입력에 대해 다른 반응 시간을 사용해야 합니다. DNA 입력이 10ng 이상인 경우 최적화를 위한 시작 시간으로 12분의 반응 시간을 선택하는 것이 좋으며 이때 생성되는 단편 크기는 주로 300-500bp 범위입니다. 사용자는 필요한 크기로 DNA 단편을 최적화하기 위해 자신의 요구 사항에 따라 2-4분 동안 DNA 단편의 길이를 늘리거나 줄일 수 있습니다.
A-2
a) 단편화 시간이 최적화되지 않은 경우 단편화된 DNA가 너무 작거나 너무 큰 경우 지침에 제공된 단편화 시간 선택 지침을 참조하여 반응 시간을 결정하고 이 시점을 대조군으로 사용하고 추가로 설정하십시오. 단편화 시간에 대한 보다 정확한 조정을 위해 3분을 연장하거나 단축하는 반응 시스템.
A-3
단편화 처리 후 DNA의 비정상적인 크기 분포
a) 단편화 시약의 해동 방법이 잘못되었거나 해동 후 시약이 완전히 혼합되지 않았습니다. 얼음 위에서 5× Fragmentation Enzyme Mix 시약을 해동합니다. 해동되면 튜브의 바닥을 가볍게 두드려 시약을 고르게 섞습니다. 시약을 소용돌이치지 마십시오!
b) DNA 입력 샘플에는 EDTA 또는 기타 오염 물질이 포함되어 있습니다. DNA 정제 단계에서 염 이온 및 킬레이트제의 고갈은 실험의 성공에 특히 중요합니다. DNA가 1xTE에 용해된 경우 지침에 제공된 방법을 사용하여 단편화를 수행합니다. 용액의 EDTA 농도가 불확실한 경우 DNA를 정제하고 후속 반응을 위해 탈이온수에 용해하는 것이 좋습니다.
c) 부정확한 초기 DNA 정량화 단편화된 DNA의 크기는 DNA 투입량과 밀접한 관련이 있습니다. 단편화 처리 전에 Qubit, Picogreen 및 기타 방법을 사용하여 DNA의 정확한 정량화가 반응 시스템에서 정확한 DNA 양을 결정하는 데 필수적입니다.
d) 반응 시스템의 준비가 지침을 따르지 않음 단편화된 반응 시스템의 준비는 지침에 따라 엄격하게 얼음 위에서 수행해야 합니다. 최상의 효과를 얻으려면 모든 반응 성분을 얼음 위에 놓고 완전히 냉각시킨 후 반응 시스템을 준비해야 합니다. 준비가 끝나면 가볍게 두드리거나 피펫으로 잘 섞어주세요. 소용돌이 하지 마십시오!
1. 잘못된 혼합 방법(와류, 격렬한 진동 등)은 라이브러리 조각의 비정상적 분포(아래 그림 참조)를 일으켜 라이브러리 품질에 영향을 미칩니다. 따라서 Fragmentation Mix 반응용액을 준비할 때 위, 아래로 부드럽게 파이펫팅하여 혼합하거나 손가락 끝을 이용하여 가볍게 두드리며 고르게 혼합하십시오. 소용돌이와 섞이지 않도록 주의하십시오.

2. 라이브러리 구축에는 고순도 DNA를 사용해야 합니다.
■ 우수한 DNA 무결성: 전기영동 밴드가 30kb 이상, 테일링 없음
■ OD260/230: >1.5
■ OD260/280: 1.7-1.9
3. DNA 투입량은 정확해야 합니다. DNA 정량은 Nanodrop 보다는 Qubit, PicoGreen 방법을 사용하는 것을 권장합니다.
4. DNA 용액에서 EDTA의 함량을 결정해야 합니다. EDTA는 단편화 반응에 큰 영향을 미칩니다. EDTA의 함량이 높으면 후속 검사 전에 DNA 정제를 수행해야 합니다.
5. 단편화 반응 용액은 얼음 위에서 준비해야 합니다. 단편화 과정은 반응 온도와 시간(특히 강화제 첨가 후)에 민감합니다. 반응 시간의 정확성을 보장하기 위해 얼음 위에서 반응 시스템을 준비하십시오.
6. 단편화 반응 시간은 정확해야 합니다. 단편화 단계의 반응 시간은 단편 산물의 크기에 직접적인 영향을 미치므로 라이브러리에서 DNA 단편의 크기 분포에 영향을 미칩니다.
1. 이 키트에 적용할 수 있는 샘플 유형은 무엇입니까?
이 키트의 적용 가능한 샘플 유형은 total RNA 또는 RNA 무결성이 우수한 정제된 mRNA일 수 있습니다. 라이브러리를 구성하기 위해 total RNA를 사용하는 경우 먼저 rRNA를 제거하기 위해 rRNA depletion kit(Cat#4992363/4992364/4992391)를 사용하는 것이 좋습니다.
2. 이 키트로 FFPE 샘플을 사용하여 라이브러리를 구성할 수 있습니까?
FFPE 샘플의 mRNA는 상대적으로 불량한 무결성으로 어느 정도 분해됩니다. 이 키트를 라이브러리 구축에 사용할 때 단편화 시간을 최적화(단편화 시간을 단축하거나 단편화를 수행하지 않음)하는 것이 좋습니다.
3. 제품 설명서에 제공된 크기 선택 단계를 사용하여 삽입된 세그먼트가 약간의 편차가 나타나는 원인은 무엇입니까?
사이즈 선택은 이 제품 매뉴얼의 사이즈 선택 단계에 따라 엄격하게 수행되어야 합니다. 편차가 있는 경우 자기 비드가 실온과 균형을 이루지 못하거나 완전히 혼합되지 않거나 피펫이 정확하지 않거나 팁에 액체가 남아 있기 때문일 수 있습니다. 실험을 위해 흡착이 적은 팁을 사용하는 것이 좋습니다.
4. 라이브러리 구축 시 어댑터 선택
라이브러리 구성 키트에는 어댑터 시약이 포함되어 있지 않으며 이 키트를 TIANSeq 단일 인덱스 어댑터(Illumina)(4992641/4992642/4992378)와 함께 사용하는 것이 좋습니다.
5. 도서관의 QC
라이브러리 정량적 검출: Qubit 및 qPCR은 각각 라이브러리의 질량 농도 및 몰 농도를 결정하는 데 사용됩니다. 작동은 제품 설명서를 엄격히 따릅니다. 라이브러리의 농도는 일반적으로 NGS 시퀀싱의 요구 사항을 충족합니다. 라이브러리 분포 범위 감지: Agilent 2100 Bioanalyzer를 사용하여 라이브러리 분포 범위 감지.
6. 증폭 주기 번호 선택
지침에 따르면 PCR 주기 수는 6-12이며 필요한 PCR 주기 수는 샘플 입력에 따라 선택해야 합니다. 고수율 라이브러리에서 과잉 증폭은 일반적으로 다양한 정도에서 발생하며, 이는 Agilent 2100 Bioanalyzer의 검출에서 표적 범위의 피크 이후 약간 더 큰 피크로 나타나거나 Qubit의 검출된 농도가 qPCR의 검출 농도보다 낮습니다. 약한 과잉 증폭은 라이브러리 시퀀싱 및 후속 데이터 분석에 영향을 미치지 않는 정상적인 현상입니다.
7. Agilent 2100 Bioanalyzer의 검출 프로필에 스파이크가 나타납니다.
Agilent 2100 Bioanalyzer 검출에서 스파이크가 나타나는 것은 샘플의 고르지 않은 단편화 때문입니다. 특정 크기의 단편이 더 많으며 이는 PCR 농축 후에 더 분명해집니다. 이 경우 크기 선택을 수행하지 않는 것이 좋습니다. 즉, 단편 분포가 작고 농축되어 균질성을 향상시킬 수 있는 15분 동안 인큐베이션 동안 단편화 조건을 94°C로 설정합니다.
제품 카테고리
왜 우리를 선택 했습니까
저희 공장은 설립 이래
품질 우선. 당사의 제품은 업계에서 우수한 평판을 얻었으며 신규 및 기존 고객 사이에서 가치 있는 신뢰를 얻었습니다.








